Создание моделей структур ДНК
С помощью программы fiber пакета 3DNA на kodomo-машине были смоделированы A-, B- и Z-формы дуплекса ДНК, последовательность одной из нитей первых двух представляет собой 5 раз повторённую последовательность "GATC", а последней - "GCGC". Полученные pdb-файлы доступны по ссылкам соответсвенно: gatc-a  ,
gatc-b
,
gatc-b  и
gcgc-z
и
gcgc-z 
Средства JMol для работы со структурами нуклеиновых кислот
Выделение различных групп атомов ДНК
Используя предопределённые множества JMol были выделены следующие группы атомов А-формы ДНК, полученной в предыдущем пункте:
- сахарофосфатный остов ДНК (рис. 1-2)

Рис.1 А-форма молекулы ДНК, вид сбоку. Серым выделены азотистые основания, красным - сахарофосфатный остов.
Рис.2 А-форма молекулы ДНК, вид по ходу цепи. Серым выделены азотистые основания, красным - сахарофосфатный остов.
Скрипт JMol для выделения сахарофосфатного остова:restrict none select all cpk 75 wireframe 40 color grey select backbone color red select backbone and *:B color translucent 0.4
- все нуклеотиды - по сути, это вся молекула ДНК (см. рис. 3)

Рис.3 А-форма молекулы ДНК, вид сбоку. Нуклеотиды раскрашены в стиле cpk. - все аденины (см. рис. 4)

Рис.4 А-форма молекулы ДНК, вид сбоку. Аденины выделены жёлтым. Команда JMol:select A - все атомы N7 во всех гуанинах (см. рис. 5)

Рис.5 А-форма молекулы ДНК, вид сбоку. Гуанины выделены серым, все атомы N7 гуанинов - зелёным и в стиле cpk. Команда JMol:select G and *.N7
Поиск файлов .pdb
На сервере RCSB Protein Data Bank были получены PDB-файлы биологических единиц молекул:
- 1GTR
 (рис. 6) - Structural basis of anticodon loop recognition by glutaminyl-tRNA synthetase (комплекс лигазы и тРНК)
(рис. 6) - Structural basis of anticodon loop recognition by glutaminyl-tRNA synthetase (комплекс лигазы и тРНК)

Рис.6 Структура комплекса тРНК и глутамил-тРНК-синтетазы, распознающей антикодон, из E.coli. - 1RH6
 (рис. 7) - Crystal structure of the excisionase-DNA complex from bacteriophage lambda (комплекс ДНК-белок)
(рис. 7) - Crystal structure of the excisionase-DNA complex from bacteriophage lambda (комплекс ДНК-белок)

Рис.7 Структура (Xis)-ДНК комплекса из бактериофага лямбда.
Проверка структур на наличие разрывов
Из файлов, полученных в предыдущем пункте, были вырезаны белки и вода, в результате чего остались только молекулы нуклеиновых кислот. Они были изучены (визуально) на наличие разрывов:
- В молекуле тРНК найден разрыв (см. рис. 8). Но при более детальном изучении выяснилось, что это отдельная молекула АТФ. Поэтому она без промедления была вырезана.

Рис.8 тРНК и АТФ из 1GTR - В молекуле ДНК разрывов не обнаружено (см. рис. 9)

Рис.8 ДНК из 1RH6
Сравнение структур 3-х форм ДНК с помощью средств JMol
Большие и малые бороздки
На поверхности парной структуры ДНК можно выделить большую и малую бороздки. Наиболее характерный вид они имеют в B-форме ДНК (рис. 11). В А-форме углубляется большая бороздка, в то время как малая бороздка становится почти незаметной (по глубине) (рис. 10). Z - форма ДНК является левозакрученной спиралью; её остов принимает зигзагообразную форму; большая бороздка едва заметна, а малая - узкая и глубокая (рис. 12)
 Рис. 10 A-форма ДНК |  Рис. 11 B-форма ДНК |  Рис. 12 Z-форма ДНК |
На примере тимина были рассмотрены, какие его атомы обращены внутрь большой бороздки, а какие - в сторону малой. Результат представлен на рис. 13, а также в файле .sk2. Положение тимина в ДНК отображено на рис. 14

Рис. 13 Положение атомов тимина относительно большой и малой бороздок B-формы ДНК
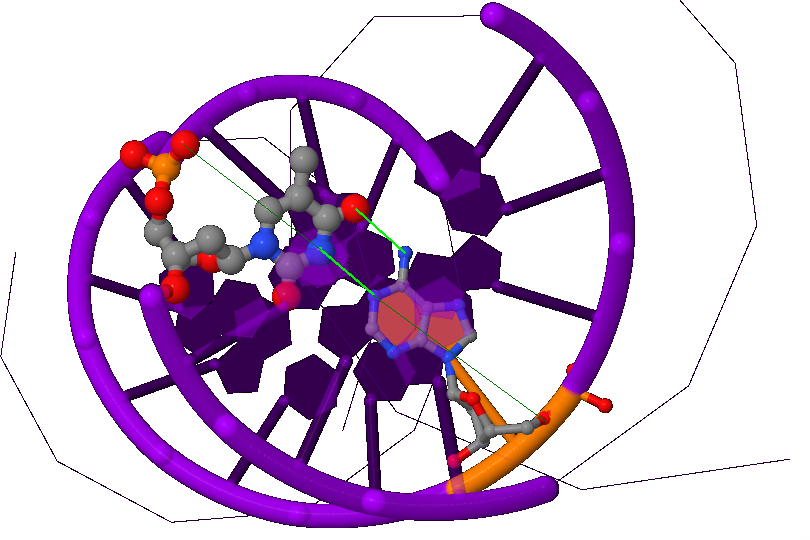
Рис.14 Тимин в B-форме ДНК.
Фиолетовым выделен остов ДНК,
серым - атомы углерода,
красным - кислорода,
синим - азота;
большим размером обозначено основание тимина,
меньшим - противолежащий аденин,
зелёным - водородная связь
Спиральные параметры ДНК
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7.98 [T]11:A - [C]24:B | 17.21 [T]11:A - [T]27:B | 15.17 [G]:11:A - [C]30:B |
| Ширина малой бороздки | 16.81 [T]11:A - [C]32:B | 11.69 [T]11:A - [A]34:B | 7.2 [G]11:A - [G]33:B |
Торсионные углы
| Форма | α | β | γ | δ | ε | ζ | χ |
|---|---|---|---|---|---|---|---|
| А-ДНК (рис. 15) | 64.1 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| В-ДНК (рис. 16) | 85.9 | 136.3 | 31.1 | 143.3 | -140.8 | -160.5 | -97.9 |
 Рис. 15 Торсионные углы T А-формы ДНК |  Рис. 16 Торсионные углы T B-формы ДНК |
Определение параметров структур НК с помощью пакета 3DNA
Торсионные углы ДНК
С помощью программ find_pair и analyze пакета 3DNA были измерены торсионные углы ДНК из 1RH6. Результаты представлены в документе Excel.
Определение структуры водородных связей
- Нуклеотиды, образующие стебли во вторичной структуре тРНК из 1GTR (# - абсолютный номер пары, № - номер нуклеотида в .pdb, N - нуклеотид):
# №_:[..N]N-----N[..N]:..№ 1 2_:[..G]G-----C[..C]:..71 | 2 3_:[..G]G-----C[..C]:..70 | 3 4_:[..G]G-----C[..C]:..69 |акцепторный 4 5_:[..G]G-----C[..C]:..68 |стебль 5 6_:[..U]U-----A[..A]:..67 | 6 7_:[..A]Ax----U[..U]:..66 | 7 49_:[..C]C-----G[..G]:..65 | 8 50_:[..G]G-----C[..C]:..64 | 9 51_:[..A]A-----U[..U]:..63 | 10 52_:[..G]G-----C[..C]:..62 |T-стебель 11 53_:[..G]G----xC[..C]:..61 | 12 54_:[..U]U-**-xA[..A]:..58 | 13 55_:[..U]Ux**+xG[..G]:..18 | 14 37_:[..A]A-*---U[..U]:..33 | 15 38_:[..U]U-*---U[..U]:..32 | 16 39_:[..U]U-----A[..A]:..31 | 17 40_:[..C]C-----G[..G]:..30 |антикодоновый 18 41_:[..C]C-----G[..G]:..29 |стебель 19 42_:[..G]G-----C[..C]:..28 | 20 43_:[..G]G-----C[..C]:..27 | 21 44_:[..C]Cx*---A[..A]:..26 | 22 10_:[..G]G-----C[..C]:..25 | 23 11_:[..C]C-----G[..G]:..24 | 24 12_:[..C]C----xG[..G]:..23 |D- 25 13_:[..A]A-**+xA[..A]:..45 |стебель 26 14_:[..A]A-*--xA[..A]:..21 | 27 15_:[..G]Gx**+xC[..C]:..48 |
Расположение указанных структур в молекуле отображено на рис.17. Примечательно, что стебли образуют спирали, напоминающую таковую в A-форме ДНК.
Рис. 17 тРНК, все подписи вынесены на рисунок - Неканонические пары оснований в структуре РНК:
Молекула содержит 8 не-Уотсон-Криковских пар оснований: 12, 13, 14, 15, 21, 25, 26, 27 предыдущего пункта.
- Дополнительные водородные связи в тРНК, стабилизирующие её тертичную структуру:
Помимо пар оснований в стеблях, есть ещё 1 изолированная пара: 19 G --- C 56
Определение возможных стекинг-взаимодействий
В файле .out (результат работы analyze) были найдены величины площади "перекрывания" колец двух последовательных пар азотистых оснований. Максимальная суммарная такая площадь в исследуемой тРНК составляет 7.11Ų (пары 20-21: GC/AC), а минимальная (не считая полного несовпадения) - 0.21Ų (пары 21-22: CG/CA).
С помощью последовательного применения программ ex_str - вырезает из staking.pdb определённую пару нуклеотидов, stack2img - pdb преобразовывает в .ps и ps2pdf - преобразовывает .ps в .pdf, были получены стандартные изображения стекинг-взаимодействия для описанных выше пар нуклеотидов; они представлены на рис. 18 и рис. 19 для максимального и минимального перекрытия соответсвенно.
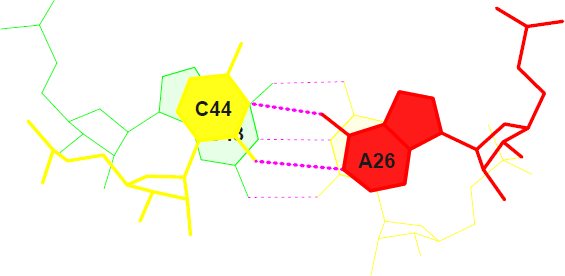
Рис. 18 Перекрытие пар 21 (сверху) и 20 (снизу)
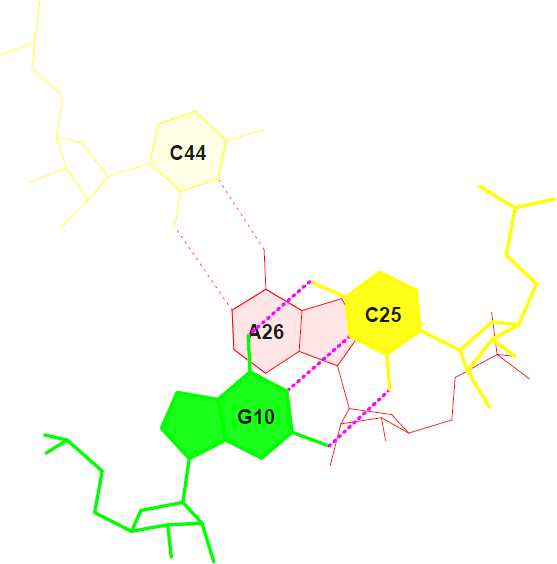
Рис. 19 Перекрытие пар 22 (сверху) и 21 (снизу)
Изображение полной структуры НК, где нуклеотиды представлены как блоки
Рис. 20 ДНК из 1RH6, представленая в виде блоков